【医療画像AI】医療AIはじめます! #001
こんにちは!こーたろーです。
大学での1年が過ぎ、漸く落ち着きました。
今年は、この技術ブログも不定期ではありますが、再度継続していきたいと考えています。
皆さんもよければゆっくり見ていってください。
大学でいろいろと教えているうちに、最近ではアプリ作成や医療AIについて勉強するようになってきました。
そこで、暫くのネタとしては、医療AIをつかってやっていきたいと思います。
今回は医療画像を使った処理を行っていきます。
画像の表示とかラベルとかを考えていきます。
環境の準備
環境は、以下のようなものを使っていきます。
・Python 3.6系
・Tensorflow 2.0.0
・matplotlib
・pillow
・imageio
・pandas
・spyder
・scikit-learn
・cython
・opencv
なお、画像データが大量にあるため、これまではGoogle Colabratoryを使用していましたが、今回はAnacondaでIPython、JupyterNotebookで行っていきます。
医療画像の入手
医療画像は、調べてみると国内外問わずオープンデータ化されているものが見つけられた。その中で、今回は、日本放射線技術学会が公開している標準ディジタル画像データベース(http://imgcom.jsrt.or.jp/minijsrtdb/)を使用してみる。
練習用データセットをダウンロードし、フォルダをセットします。
今回は、pythonファイルと同様の階層にフォルダをセットしています。
ライブラリ、オブジェクトのインポート
今回使うライブラリの全てをインポートします。
import numpy as np import cv2 import os from tensorflow.python.keras.utils import to_categorical from matplotlib import pyplot as plt from os import listdir from keras.preprocessing.image import load_img, img_to_array
データのロード
医療画像では、例えばCT画像であれば、その画像に対して画像に病気となっている箇所があるかないかの正解ラベルのようなものを準備して、機械学習をかけていきます。今回は、画像データに対して、標準ディジタル画像データベースで公開されているデータを用いて、画像に対する正解ラベルが設定されているので、それを読み込む。
gs = np.loadtxt('result.csv', delimiter=',' , skiprows=1, usecols=(1)) gs = to_categorical(gs,2)
読み込んだあとは、正解ラベルをダミー化して、0,1のラベルにしておく。
csvファイルは、画像ファイルのファイル名および画像に対する異状の有無の2つのカラムになっており、2列目が「nodule(病巣の塊)」の有無を表している。
ダミー化を行うと、以下のような結果になる。

なお、画像のラベルについては、以下のようにして取り出してる。
fname = np.genfromtxt('result.csv',dtype='str', delimiter=',', skip_header=1, usecols=(0))
結果としては、

のように、画像の名称(ファイル名)が登録されている。
画像ファイルの読み込みと表示
画像ファイルは、前にダウンロードした練習用データセットを展開すると、「PNG」形式のフォルダがでてくる。今回は、このPNGファイルで読み込みと表示を行う。
img01=cv2.imread("./chestimage_PNG/JPCLN001.png")
plt.imshow(img01)
plt.show()
OpenCVのimredメソッドを使って、フォルダ内の画像を指定し表示させる。
表示画像は、サンプルの1つをピックアップしている。
表示結果としては、次のようになる。
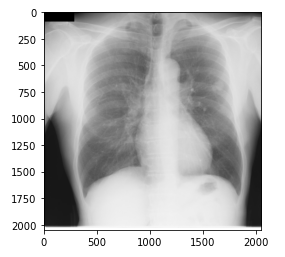
画像データを取り扱っている人ならご承知のとおり、このimredメソッドは、各ビットの画素を配列で処理している。
つまり、img01自体は、配列となっている。
画像をnp配列に変換するには、以下のソースで行う。img_to_arrayによって配列変換し、numpyで扱えるようにする。
img01-32 = np.array(img_to_array(load_img("./chestimage_PNG/JPCLN001.png",target_size=(32,32))))/255.0
同一フォルダ内に保存されてる全画像データの取得
ダウンロードした画像ファイルは、「chestimages_PNG」に格納されている。それらのファイル名を取得する。なお、本ファイルには、隠しファイルとして「.」から始まるファイルが存在しているため、それは読み込まないようにする。
files_list_PNG = sorted([filename for filename in listdir('./chestimage_PNG/') if not filename.startswith('.')]) img_PNG_ALL = np.zeros((len(files_PNG), 32, 32, 3)) for i in range(0,len(files_PNG)): filename ='./chestimage_PNG/%s' % files_PNG[i] chest_PNG_ALL[i]=np.array(img_to_array(load_img(filename,target_size=(32,32))))/255.0
ダウンロードしたファイルは、10個あり、それぞれを32×32ビット3チャンネルの画像として、全てをimg_PNG_ALLに配列で取り込む。
次回は、DICOM画像について取り扱っていきます。
少し癖があるようですので、やり方を勉強しておきます。
データセットのダウンロードの仕方も説明できればと思います。
ではでは。